SAM-TB
首页
我的数据
我的分析
我的样本
帮助中心
FAQ
English
登录
##一线药物敏感性和耐药性预测 ###耐药突变SNV检测阈值 在耐药突变的鉴定中,SNV需满足以下条件:测序深度至少为5×,突变碱基的频率不小于10%,并且正反至少各一条reads支持突变碱基。 ###四种一线药物(异烟肼,利福平,乙胺丁醇,吡嗪酰胺)耐药突变位点数据库 基于从参考文献[1]和[2]中提取的耐药决定突变位点数据库进行异烟肼,利福平,乙胺丁醇,吡嗪酰胺耐药突变位点检测。该数据库中,异烟肼耐药相关基因包括inhA、katG、ahpC、fabG1、kasA,利福平耐药相关基因包括rpoB和rpoC,乙胺丁醇耐药相关基因包括embA、embB、embC,和吡嗪酰胺耐药相关基因包括pncA、panD和rpsA。 ###四种药物耐药以及敏感性预测原理 对这四种药物敏感性的预测则是根据参考文献[1]实现的。如图所示,利用菌株的测序数据,并结合耐药决定突变以及无害突变数据库,判断菌株对四种药物中的每一种药物是否耐药,结果包括耐药,敏感以及未检测到已知耐药突变。 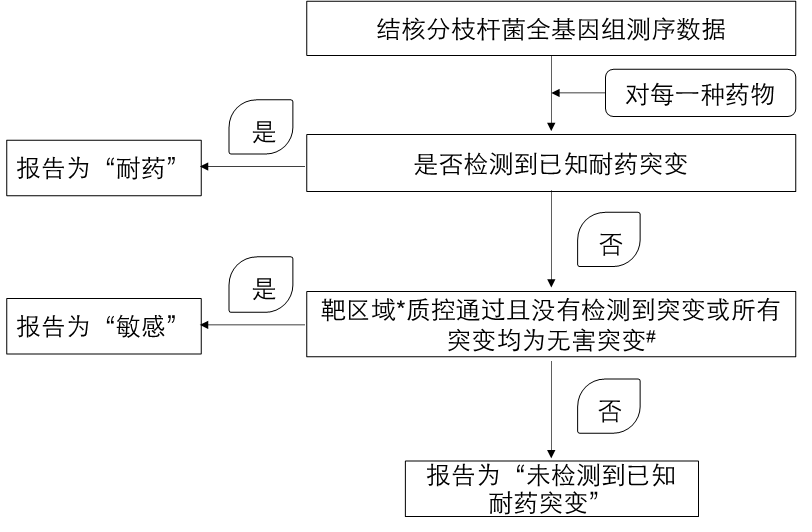 注:* 靶区域是与四种一线药物耐药相关的基因和启动子区域,包括katG, inhA, fabG1, ahpC, rpoB, embA, embB, embC, pncA。#无害突变被认为对菌株耐药性没有影响。 ###适用范围和局限性 (1)由于当前技术和数据库版本的局限性,本系统分析结果仅可作为科学研究的参考,不应作为临床诊断标准; (2)本系统分析结果仅可作为临床治疗提示,不应作为治疗依据,本系统不对用户依据分析结果采取的任何行为所造成的后果(包括精神、经济和法律方面)负责; (3)随着对耐药机制研究的深入,一些突变的耐药性或敏感性可能会发生改变,网站会定期进行更新。 ###更新日志 ##### 2024.04 2023年11月,世界卫生组织(World Health Organization, WHO)发布了第二版《结核分枝杆菌耐药相关基因突变目录》[3],基于这份《目录》,对SAM-TB的耐药突变位点数据库进行了更新和扩充。表1显示新版数据库中四种一线药物的耐药相关基因,表2及表3分别显示新版数据库中调整的耐药和敏感突变位点的数量信息。 表1: 新版数据库中,四种一线药物的耐药相关基因 |药物|候选耐药基因| |----|----| |异烟肼|ahpC,fabG1,inhA,kasA,katG| |利福平|rpoB,rpoC| |吡嗪酰胺|pncA,panD,rpsA| |乙胺丁醇|embA,embB,embC,embR| 表2: 新版数据库中,修改及新增的耐药突变位点数量 |药物|No change|Down from High to Low|Up from sensitive to Low|Up from sensitive to High|Up from Low to High|New Low|New High| |----|----|----|----|----|----|----|----| |异烟肼|329|2|-|-|3|20|2| |利福平|7751|8|1|-|6|56|2| |吡嗪酰胺|445|40|1|1|41|25|13| |乙胺丁醇|190|-|-|-|5|-|-| 注:在新版数据库中,耐药突变的可信度变化调整如下所示。 - “No change”:耐药突变的可信度保持不变; - “Down from High to Low”:耐药突变的可信度从“高可信度(High-confidence)”调整为“低可信度(Low-confidence)”; - “Up from sensitive to Low”:原本标记为“敏感”的突变现调整为“低可信度”的耐药突变; - “Up from sensitive to High” :原本标记为“敏感”的突变现调整为“高可信度”的耐药突变; - “Up from Low to High”:耐药突变的可信度从“低可信度”提升至“高可信度”; - “New Low”:新增的“低可信度”耐药突变; - “New High”:新增的“高可信度”耐药突变。 表3: 新版数据库中,修改及新增的敏感突变位点数量 |药物|No change|Down from Low to sensitive|New sensitive| |----|----|----|----| |异烟肼|1558|-|1225| |利福平|1232|2|1415| |吡嗪酰胺|214|3|614| |乙胺丁醇|3601|-|462| 注:在新版数据库中,敏感突变的变化调整如下所示。 - “No change”:敏感突变的敏感性保持不变; - “Down from Low to sensitive”:原本标记为“低可信度”的耐药突变现调整为“敏感突变”; - “New sensitive”:新增的“敏感突变”。 ###参考文献 [1] Walker TM, Kohl TA, Omar SV, Hedge J, Del Ojo Elias C, Bradley P, et al. Whole-genome sequencing for prediction of Mycobacterium tuberculosis drug susceptibility and resistance: a retrospective cohort study. The Lancet Infectious diseases. 2015;15(10):1193-202. doi: 10.1016/S1473-3099(15)00062-6. PubMed PMID: 26116186; PubMed Central PMCID: PMC4579482. [2] Jody E Phelan, Denise M O'Sullivan, Diana Machado, Jorge Ramos, Yaa E A Oppong, Susana Campino, Justin O'Grady, Ruth McNerney, Martin L Hibberd, Miguel Viveiros, Jim F Huggett, Taane G Clark. Integrating informatics tools and portable sequencing technology for rapid detection of resistance to anti-tuberculous drugs. Genome Medicine. 2019;11(1):41. doi: 10.1186/s13073-019-0650-x. PubMed PMID: 31234910; PubMed Central PMCID: PMC6591855. [3] WHO. Catalogue of mutations in Mycobacterium tuberculosis complex and their association with drug resistance, 2nd edn. 2023. https://www.who.int/publications/i/item/9789240082410.
<< 返回帮助中心
标题:
描述:
感谢您使用我们的服务,我们将尽快邮件回复您。